17 Quelques modules d'intérêt en bioinformatique¶
Nous allons aborder dans ce chapitre quelques modules très importants en bioinformatique. Le premier NumPy permet notamment de manipuler des vecteurs et des matrices. Le module Biopython permet de travailler sur des données biologiques, comme des séquences (nucléiques et protéiques) ou des structures (fichiers PDB). Le module matplotlib permet de créer des graphiques depuis Python. Enfin, le module pandas est très performant pour l'analyse de données, et scipy étend les possibilités offertes par NumPy, notamment en proposant des algorithmes couramment utilisés en calcul scientifique.
Ces modules ne sont pas fournis avec la distribution Python de base (contrairement à tous les autres modules vus précédemment). Avec la distribution Miniconda que nous vous avons conseillé d'utiliser (consultez pour cela la documentation en ligne), vous pouvez rapidement les installer avec la commande :
Dans ce chapitre, nous vous montrerons quelques exemples d’utilisation de ces modules pour vous convaincre de leur pertinence.
17.1 Module NumPy¶
Le module NumPy est incontournable en bioinformatique. Il permet d'effectuer des calculs sur des vecteurs ou des matrices, élément par élément, via un nouveau type d'objet appelé array.
On charge le module NumPy avec la commande :
On utilise très souvent un nom raccourci pour NumPy :
17.1.1 Objets de type array¶
Les objets de type array correspondent à des tableaux à une ou plusieurs dimensions et permettent d'effectuer du calcul vectoriel. La fonction array() convertit un container (comme une liste ou un tuple) en un objet de type array. Voici un exemple simple de conversion d'une liste à une dimension en objet array :
>>> import numpy as np
>>> a = [1, 2, 3]
>>> np.array(a)
array([1, 2, 3])
>>> b = np.array(a)
>>> b
array([1, 2, 3])
>>> type(b)
<type 'numpy.ndarray'>
Nous avons converti la liste [1, 2, 3] en array. Nous aurions obtenu le même résultat si nous avions converti le tuple (1, 2, 3) en array.
Par ailleurs, lorsqu'on demande à Python d'afficher le contenu d'un objet array, le mot array et les symboles ([ et ]) sont utilisés pour le distinguer d'une liste (délimitée par les caractères [ et ]) ou d'un tuple (délimité par les caractères ( et )).
Un objet array ne contient que des données homogènes, c'est-à-dire d'un type identique.
Il est possible de créer un objet array à partir d'une liste contenant des entiers et des chaînes de caractères, mais dans ce cas, toutes les valeurs seront comprises par NumPy comme des chaînes de caractères :
>>> a = np.array([1, 2, "tigre"])
>>> a
array(['1', '2', 'tigre'], dtype='<U21')
>>> type(a)
<class 'numpy.ndarray'>
De même, il est possible de créer un objet array à partir d'une liste constituée d'entiers et de floats, mais toutes les valeurs seront alors comprises par NumPy comme des floats :
Ici, la notation 1. indique qu'il s'agit du float 1.0000... et pas de l'entier 1.
Sur un modèle similaire à la fonction range(), la fonction arange() permet de construire un array à une dimension de manière simple.
Comme avec range(), on peut spécifier en argument une borne de début, une borne de fin et un pas :
Un autre avantage de la fonction arange() est qu'elle génère des objets array qui contiennent des entiers ou des floats selon l'argument qu'on lui passe :
>>> np.arange(10)
array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])
>>> np.arange(10.0)
array([ 0., 1., 2., 3., 4., 5., 6., 7., 8., 9.])
La différence fondamentale entre un objet array à une dimension et une liste (ou un tuple) est que celui-ci est considéré comme un vecteur. Par conséquent, on peut effectuer des opérations élément par élément sur ce type d'objet, ce qui est bien commode lorsqu'on analyse de grandes quantités de données. Regardez ces exemples :
>>> v = np.arange(4)
>>> v
array([0, 1, 2, 3])
>>> v + 1
array([1, 2, 3, 4])
>>> v + 0.1
array([ 0.1, 1.1, 2.1, 3.1])
>>> v * 2
array([0, 2, 4, 6])
>>> v * v
array([0, 1, 4, 9])
Avec les listes, ces opérations n'auraient été possibles qu'en utilisant des boucles. Nous vous encourageons donc à utiliser dorénavant les objets array lorsque vous aurez besoin de faire des opérations élément par élément.
Notez également que, dans le dernier exemple de multiplication (ligne 10), l'array final correspond à la multiplication élément par élément des deux arrays initiaux.
17.1.2 Array et dimensions¶
Il est aussi possible de construire des objets arrays à deux dimensions, il suffit de passer en argument une liste de listes à la fonction array() :
On peut aussi créer des tableaux à trois dimensions en passant comme argument à la fonction array() une liste de listes de listes :
>>> x = np.array([[[1, 2], [2, 3]], [[4, 5], [5, 6]]])
>>> x
array([[[1, 2],
[2, 3]],
[[4, 5],
[5, 6]]])
La fonction array() peut créer des tableaux à n'importe quel nombre de dimensions. Toutefois ça devient vite compliqué lorsqu'on dépasse trois dimensions. Retenez qu'un objet array à une dimension peut être assimilé à un vecteur et un array à deux dimensions à une matrice. Nous nous focaliserons dans la suite sur des arrays 1D ou 2D.
Avant de continuer, il est important de définir comment sont organisés ces arrays 2D qui représentent des matrices. Il s'agit de tableaux de nombres qui sont organisés en lignes et en colonnes comme le montre la figure 1. Les indices indiqués dans cette figure seront définis un peu plus loin dans la rubrique Indices.
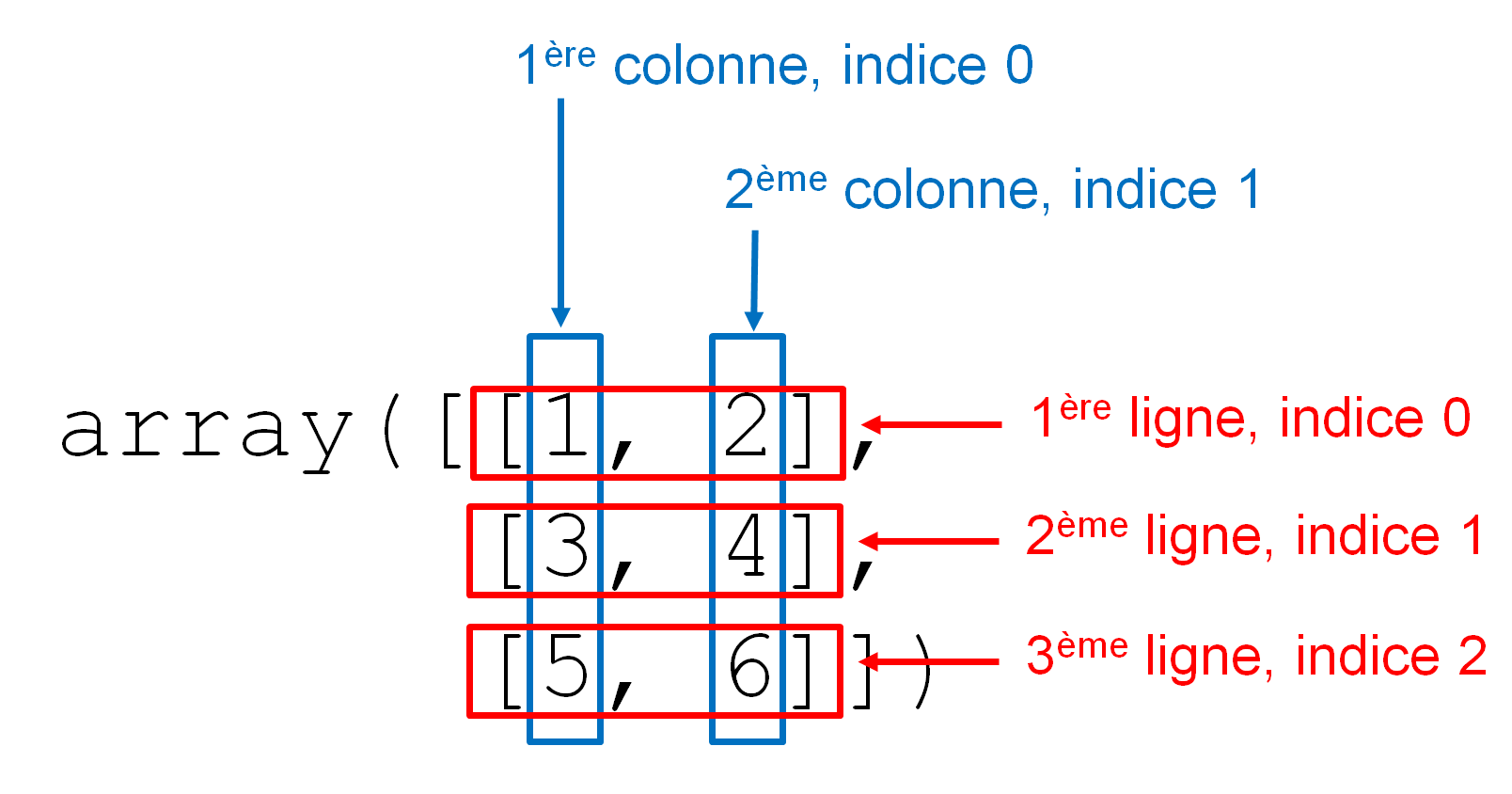
Voici quelques attributs intéressants pour décrire un objet array :
.ndimrenvoie le nombre de dimensions (par exemple, 1 pour un vecteur et 2 pour une matrice)..shaperenvoie les dimensions sous forme d'un tuple. Dans le cas d'une matrice (array à deux dimensions), la première valeur du tuple correspond au nombre de lignes et la seconde au nombre de colonnes..sizerenvoie le nombre total d'éléments contenus dans l'array.
>>> v = np.arange(4)
>>> v
array([0, 1, 2, 3])
>>> v.ndim
1
>>> v.shape
(4,)
>>> v.size
4
>>> w = np.array([[1, 2], [3, 4], [5, 6]])
>>> w
array([[1, 2],
[3, 4],
[5, 6]])
>>> w.ndim
2
>>> w.shape
(3, 2)
>>> w.size
6
Et la méthode .reshape() renvoie un nouvel array avec les dimensions spécifiées :
>>> a = np.arange(0, 6)
>>> a
array([0, 1, 2, 3, 4, 5])
>>> a.shape
(6,)
>>> b = a.reshape((2, 3))
>>> b
array([[0, 1, 2],
[3, 4, 5]])
>>> b.shape
(2, 3)
>>> a
array([0, 1, 2, 3, 4, 5])
Notez bien que le array a n'a pas été modifié et que a.reshape((2, 3)) n'est pas la même chose que a.reshape((3, 2)) :
La méthode .reshape() attend que les nouvelles dimensions soient compatibles avec la dimension initiale de l'objet array, c'est-à-dire que le nombre d'éléments contenus dans les différents arrays soit le même.
Dans nos exemples précédents, \(6 = 2 \times 3 = 3 \times 2\).
Si les nouvelles dimensions ne sont pas compatibles avec les dimensions initiales, la méthode .reshape() génère une erreur.
>>> a = np.arange(0, 6)
>>> a
array([0, 1, 2, 3, 4, 5])
>>> a.shape
(6,)
>>> d = a.reshape((3, 4))
Traceback (most recent call last):
File "<stdin>", line 1, in <module>
ValueError: cannot reshape array of size 6 into shape (3,4)
La méthode .resize() par contre ne déclenche pas d'erreur dans une telle situation et ajoute des 0 jusqu'à ce que le nouvel array soit rempli, ou bien coupe la liste initiale.
>>> a = np.arange(0, 6)
>>> a.shape
(6,)
>>> a.resize((3, 3), refcheck=False)
>>> a.shape
(3, 3)
>>> a
array([[0, 1, 2],
[3, 4, 5],
[0, 0, 0]])
>>> b = np.arange(0, 10)
>>> b.shape
(10,)
>>> b.resize((2, 3), refcheck=False)
>>> b.shape
(2, 3)
>>> b
array([[0, 1, 2],
[3, 4, 5]])
Attention, cette modification de la forme de l'array par la méthode .resize() est faite « sur place » (in place), c'est-à-dire que la méthode ne renvoie rien mais l'array est bel et bien modifié (à l'image des méthodes sur les listes comme .reverse(), cf. chapitre 11 Plus sur les listes). Si l'option refcheck=False n'est pas présente, Python peut parfois renvoyer une erreur s'il existe des références vers l'array qu'on souhaite modifier.
Il existe aussi la fonction np.resize() qui, dans le cas d'un nouvel array plus grand que l'array initial, va répéter l'array initial afin de remplir les cases manquantes :
>>> a = np.arange(0, 6)
>>> a.shape
(6,)
>>> c = np.resize(a, (3, 5))
>>> c.shape
(3, 5)
>>> c
array([[0, 1, 2, 3, 4],
[5, 0, 1, 2, 3],
[4, 5, 0, 1, 2]])
>>> a
array([0, 1, 2, 3, 4, 5])
Notez que cette fonction np.resize() renvoie un nouvel array mais ne modifie pas l'array initial contrairement à la méthode .resize() décrite ci-dessus.
Depuis le début de ce chapitre, nous avons toujours montré l'affichage d'un array dans l'interpréteur :
>>> a = np.array(range(10))
>>> a
array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])
>>> a2 = np.ones((3, 3))
>>> a2
array([[1., 1., 1.],
[1., 1., 1.],
[1., 1., 1.]])
Nous avons déjà indiqué que Python affiche systématiquement le mot array ainsi que les parenthèses, crochets et virgules pour séparer les éléments. Attention toutefois si vous utilisez la fonction print() car l'affichage est différent. Le mot array, les parenthèses et les virgules disparaissent :
[0 1 2 3 4 5 6 7 8 9] et une liste contenant les même éléments [0, 1, 2, 3, 4, 5, 6, 7, 8, 9]. Dans ce cas, seule la présence ou l'absence de virgules permet de trancher s'il s'agit d'un array ou d'une liste.
17.1.3 Méthodes de calcul sur les arrays et l'argument axis¶
Chaque array NumPy qui est créé possède une multitude de méthodes. Nombre d'entre elles permettent de faire des calculs de base comme .mean() pour la moyenne, .sum() pour la somme, .std() pour l'écart-type, .max() pour extraire le maximum, .min() pour extraire le minimum, etc. La liste exhaustive est ici. Par défaut, chacune de ces méthodes effectuera l'opération sur l'array entier, quelle que soit sa dimensionnalité. Par exemple :
>>> import random as rd
>>> l = list(range(8))
>>> rd.shuffle(l)
>>> l
[2, 7, 6, 4, 0, 3, 1, 5]
>>> a = np.resize(l, (4, 2))
>>> a
array([[2, 7],
[6, 4],
[0, 3],
[1, 5]])
>>> a.max()
7
La méthode .max() nous a bien renvoyé la valeur maximale 7. Un argument très utile existant dans toutes ces méthodes est axis. Pour un array 2D, axis=0 signifie qu'on fera l'opération le long de l'axe 0, à savoir les lignes. C'est-à-dire que l'opération se fait en faisant varier les lignes. On récupère ainsi une valeur par colonne :
Dans l'array 1D récupéré, le premier élément vaut 6 (maximum de la 1ère colonne) et le second vaut 7 (maximum de la seconde colonne).
Avec axis=1 on fait une opération similaire mais en faisant varier les colonnes. On récupère ainsi une valeur par ligne :
L'array 1D récupéré a 4 éléments correspondant au maximum de chaque ligne.
On comprend la puissance de cet argument axis. À nouveau, il est possible, en une ligne, de faire des calculs qui pourraient être très fastidieux avec les listes traditionnelles.
17.1.4 Indices¶
Pour récupérer un ou plusieurs élément(s) d'un objet array, vous pouvez utiliser les indices ou les tranches, de la même manière qu'avec les listes :
>>> a = np.arange(10)
>>> a
array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])
>>> a[5:]
array([5, 6, 7, 8, 9])
>>> a[::2]
array([0, 2, 4, 6, 8])
>>> a[1]
1
>>> a = np.array([[1, 2], [3, 4]])
>>> a
array([[1, 2],
[3, 4]])
>>> a[:,0]
array([1, 3])
>>> a[0,:]
array([1, 2])
>>> a[1, 1]
4
Lignes 5 à 8. La syntaxe a[i,:] renvoie la ligne d'indice i, et a[:,j] renvoie la colonne d'indice j. Les tranches sont évidemment aussi utilisables sur un array à deux dimensions.
Lignes 9 à 10. La syntaxe a[i, j] renvoie l'élément à la ligne d'indice i et à la colonne d'indice j. Notez que NumPy suit la convention mathématiques des matrices, à savoir, on définit toujours un élément par sa ligne puis par sa colonne. En mathématiques, l'élément \(a_{ij}\) d'une matrice \(A\) se trouve à la \(i^{ème}\) ligne et à la \(j^{ème}\) colonne.
- Pour un array 2D, si un seul indice est donné, par exemple
a[i], on récupère la ligne d'indiceisous forme d'array 1D :
- Pour cette raison, la syntaxe
a[i][j]est également valide pour récupérer un élément :
Bien que cela soit possible, nous vous recommandons tout de même la syntaxe a[i, j] qui est plus proche de la définition mathématiques d'un élément de matrice.
17.1.5 Copie d'arrays¶
Comme pour les listes, nous attirons votre attention sur la copie d'arrays :
>>> a = np.arange(5)
>>> a
array([0, 1, 2, 3, 4])
>>> b = a
>>> b[2] = -300
>>> b
array([ 0, 1, -300, 3, 4])
>>> a
array([ 0, 1, -300, 3, 4])
Par défaut la copie d'arrays se fait par référence comme pour tous les containers en Python (listes, tuples, dictionnaires, etc.).
Afin d'éviter le problème, vous pouvez soit utiliser la fonction np.array() qui crée une nouvelle copie distincte de l'array initial, soit la fonction copy.deepcopy() comme pour les listes (cf. chapitre 11 Plus sur les listes) :
>>> a = np.full((2, 2), 0)
>>> a
array([[0, 0],
[0, 0]])
>>> b = np.array(a)
>>> b[1, 1] = -300
>>> c = copy.deepcopy(a)
>>> c[1, 1] = -500
>>> a
array([[0, 0],
[0, 0]])
>>> b
array([[ 0, 0],
[ 0, -300]])
>>> c
array([[ 0, 0],
[ 0, -500]])
La fonction np.full() est expliquée dans la rubrique suivante.
On pourra noter que la stratégie b = np.array(a) fait bien une copie distincte de l'array a quelle que soit sa dimensionnalité. Ceci n'était pas le cas avec la fonction list() pour les copies de listes à partir de la dimensionnalité 2 :
17.1.6 Construction automatique de matrices¶
Il peut être parfois pénible de construire une matrice (array à deux dimensions) à l'aide d'une liste de listes. Le module NumPy possède quelques fonctions pratiques pour initialiser des matrices. Par exemple, Les fonctions zeros() et ones() construisent des objets array contenant des 0 ou des 1. Il suffit de leur passer en argument un tuple indiquant les dimensions voulues.
>>> np.zeros((2, 3))
array([[0., 0., 0.],
[0., 0., 0.]])
>>> np.ones((3, 3))
array([[1., 1., 1.],
[1., 1., 1.],
[1., 1., 1.]])
Par défaut, les fonctions zeros() et ones() génèrent des floats, mais vous pouvez demander des entiers en passant le type (par exemple int, float, etc.) en second argument :
Enfin, si vous voulez construire une matrice avec autre chose que des 0 ou des 1, vous avez à votre disposition la fonction full() :
>>> np.full((2, 3), 7, int)
array([[7, 7, 7],
[7, 7, 7]])
>>> np.full((2, 3), 7, float)
array([[ 7., 7., 7.],
[ 7., 7., 7.]])
Nous construisons ainsi une matrice constituée de 2 lignes et 3 colonnes. Celle-ci ne contient que le chiffre 7 sous formes d'entiers (int) dans le premier cas et de floats dans le second.
Le module numpy contient aussi des fonctions pour lire des données à partir de fichiers et créer des arrays automatiquement. Cela se révèle bien pratique car la plupart du temps les données que l'on analyse proviennent de fichiers. La fonction la plus simple à prendre en main est np.loadtxt(). Celle-ci lit un fichier organisé en lignes / colonnes. Par exemple, imaginons que nous ayons un fichier donnees.dat contenant :
La fonction prend en argument le nom du fichier et renvoie un array 2D directement :
Pratique, non ? Attention toutefois aux points suivants :
- chaque ligne doit avoir le même nombre de colonnes, la fonction ne gère pas les données manquantes ;
- chaque donnée est convertie en float, donc si une chaîne est rencontrée la fonction renvoie une erreur ;
- par défaut, les données doivent être séparées par n'importe quelle combinaison d'espace(s) et/ou de tabulations.
Nous vous conseillons vivement de consulter la documentation complète de cette fonction. np.loadtxt() contient tout un tas d'arguments par mot-clé permettant de récupérer telles ou telles lignes / colonnes, ignorer des lignes de commentaire, changer le séparateur par défaut (par exemple la virgule , pour les fichiers .csv), etc., qui peuvent se révéler commodes.
L'opération inverse consistant à sauver un array dans un fichier se fait avec la fonction np.savetxt() :
>>> a = np.reshape(range(1, 10), (3, 3))
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> np.savetxt("out.dat", a)
Ceci générera le fichier out.dat contenant les lignes suivantes :
1.000000000000000000e+00 2.000000000000000000e+00 3.000000000000000000e+00
4.000000000000000000e+00 5.000000000000000000e+00 6.000000000000000000e+00
7.000000000000000000e+00 8.000000000000000000e+00 9.000000000000000000e+00
On voit que la fonction écrit par défaut les données comme des floats en notation scientifique. Bien sûr il existe de nombreuses options possibles permettant de changer le format, les séparateurs, etc.
Il existe d'autres fonctions plus avancées telles que np.genfromttxt() gérant les données manquantes, ou encore np.load() et np.fromfile() permettant de lire des données au format binaire. De même, il existe des fonctions ou méthodes permettant d'écrire au format binaire : np.save() ou .tofile(). Le format binaire possède en général l'extension .npy ou .npz lorsque les données sont compressées. L'avantage d'écrire au format binaire est que cela prend moins de place pour les gros tableaux.
17.1.7 Concaténation d'arrays¶
Il peut être très utile de concaténer un ou plusieurs arrays. Il existe pour cela plusieurs fonctions dans NumPy, nous développerons celle qui nous parait la plus intuitive et directe : np.concatenate().
Pour les arrays 1D, np.concatenate() prend en argument un tuple contenant les arrays à concaténer :
>>> a1 = np.array((0, 1))
>>> a2 = np.array((3, 4))
>>> a1
array([0, 1])
>>> a2
array([3, 4])
>>> np.concatenate((a1, a2))
array([0, 1, 3, 4])
>>> np.concatenate((a2, a1))
array([3, 4, 0, 1])
>>> np.concatenate((a1, a2, a1, a2))
array([0, 1, 3, 4, 0, 1, 3, 4])
Pour les arrays 2D ça se complique un peu car on peut concaténer des lignes ou des colonnes ! Ainsi, np.concatenate() prend un argument optionnel, à savoir axis. Comme nous l'avions expliqué plus haut, celui-ci va indiquer à NumPy si on veut concaténer le long de l'axe 0 (les lignes) ou le long de l'axe 1 (les colonnes). Voyons un exemple :
>>> a1 = np.reshape(np.array(range(6)), (3, 2))
>>> a2 = a * 5
>>> a1
array([[0, 1],
[2, 3],
[4, 5]])
>>> a2
array([[ 0, 5],
[10, 15],
[20, 25]])
>>>
>>> np.concatenate((a1, a2), axis=0)
array([[ 0, 1],
[ 2, 3],
[ 4, 5],
[ 0, 5],
[10, 15],
[20, 25]])
>>> np.concatenate((a1, a2), axis=1)
array([[ 0, 1, 0, 5],
[ 2, 3, 10, 15],
[ 4, 5, 20, 25]])
En ligne 12, on concatène par ligne (axis=0), c'est-à-dire qu'on ajoute les lignes du deuxième array a2 à celles de l'array a1. En ligne 19, on concatène par colonne (axis=1). Attention, il vaut bien veiller à ce que la concaténation soit possible en terme de dimensionalité. Par exemple, en ligne 12, il faut que les 2 arrays a1 et a2 aient le même nombre de colonnes.
Ces opérations de concaténation sont très importantes. On les utilise par exemple si on a des données dans plusieurs fichiers différents et qu'on veut obtenir un array unique au final. On verra qu'on peut faire le même genre de chose avec les fameux dataframes du module pandas. Lisez bien également les recommandations dans la dernière rubrique 17.1.10 Quelques conseils sur quand utiliser la concaténation d'arrays NumPy.
17.1.8 Un peu d'algèbre linéaire¶
Après avoir manipulé les objets array comme des vecteurs et des matrices, voici quelques fonctions pour faire de l'algèbre linéaire.
La fonction transpose() renvoie la transposée d'un array. Par exemple, pour une matrice :
>>> a = np.resize(np.arange(1, 10), (3, 3))
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> np.transpose(a)
array([[1, 4, 7],
[2, 5, 8],
[3, 6, 9]])
Tout objet array possède un attribut .T qui contient la transposée, il est ainsi possible d'utiliser cette notation objet plus compacte :
La fonction dot() permet de multiplier deux matrices.
>>> a = np.resize(np.arange(4), (2, 2))
>>> a
array([[0, 1],
[2, 3]])
>>> np.dot(a, a)
array([[ 2, 3],
[ 6, 11]])
>>> a * a
array([[0, 1],
[4, 9]])
Notez bien que dot(a, a) renvoie le produit matriciel entre deux matrices, alors que a * a renvoie le produit élément par élément.
Dans le module NumPy, il existe également des objets de type matrix pour lesquels les multiplications de matrices sont différents, mais nous ne les aborderons pas ici.
Pour toutes les opérations suivantes, nous utiliserons des fonctions du sous-module linalg de NumPy. La fonction inv() renvoie l'inverse d'une matrice carrée, det() son déterminant et eig() ses vecteurs et valeurs propres.
>>> a = np.diag((1, 2, 3))
>>> a
array([[1, 0, 0],
[0, 2, 0],
[0, 0, 3]])
>>> np.linalg.inv(a)
array([[1. , 0. , 0. ],
[0. , 0.5 , 0. ],
[0. , 0. , 0.33333333]])
>>> np.linalg.det(a)
6.0
>>> np.linalg.eig(a)
(array([1., 2., 3.]), array([[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]]))
Ligne 1. La fonction .diag() permet de générer une matrice diagonale.
Lignes 12 à 15. La fonction eig() renvoie un tuple dont le premier élément (d'indice 0) correspond aux valeurs propres et le second (d'indice 1) aux vecteurs propres. Une façon commode de récupérer ces éléments est d'utiliser cette fonction avec l'affectation multiple :
>>> eigvals, eigvecs = np.linalg.eig(a)
>>> eigvals
array([1., 2., 3.])
>>> eigvecs
array([[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]])
>>> eigvecs[0]
array([1., 0., 0.])
>>> eigvecs[1]
array([0., 1., 0.])
>>> eigvecs[2]
array([0., 0., 1.])
Lignes 2 et 3. eigvals est un array 1D contenant les 3 valeurs propres.
Lignes 4 à 13. eigvecs est un array 2D contenant les 3 vecteurs propres (un par ligne), que l'on peut récupérer avec eigvecs[0], eigvecs[1] et eigvecs[2].
17.1.9 Parcours de matrice et affectation de lignes / colonnes¶
Lorqu'on a une matrice, on est souvent amené à la parcourir par ligne ou par colonne. Une fonctionnalité bien commode vient du fait que les arrays NumPy sont directement itérables par ligne :
>>> a = np.reshape(np.arange(1, 10), (3, 3))
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> for row in a:
... print(row, type(row))
...
[1 2 3] <class 'numpy.ndarray'>
[4 5 6] <class 'numpy.ndarray'>
[7 8 9] <class 'numpy.ndarray'>
À chaque itération, la variable row est un array 1D correspondant à chaque ligne de a. Cela est lié au fait que l'utilisation d'un indiçage unique a[i] pour un array 2D correspond à sa ligne d'indice i (cf. rubrique Indices ci-dessus).
Pour itérer sur les colonnes, on pourra utiliser l'astuce d'itérer sur la transposée de l'array a, c'est-à-dire a.T :
>>> for col in a.T:
... print(col, type(col))
...
[1 4 7] <class 'numpy.ndarray'>
[2 5 8] <class 'numpy.ndarray'>
[3 6 9] <class 'numpy.ndarray'>
À chaque itération, la variable col est un array 1D correspondant à chaque colonne de a.
On se souvient de l'affectation multiple x, y = 1, 2 qui permettait d'affecter des valeurs à plusieurs variables à la fois. Et bien, il est possible d'utiliser cette fonctionnalité aussi avec les arrays NumPy :
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> l1, l2, l3 = a
>>> l1
array([1, 2, 3])
>>> l2
array([4, 5, 6])
>>> l3
array([7, 8, 9])
Par défaut, cela se fait sur les lignes de l'array 2D. Cette fonctionnalité provient à nouveau du fait que pour NumPy a[i] correspond à la ligne d'indice i d'un array 2D.
Pour utiliser l'affectation multiple sur les colonnes, il suffit d'utiliser la transposée a.T :
17.1.10 Masques booléens¶
Une des grandes puissance des arrays NumPy est qu'ils supportent les masques booléens. Avant de les définir, il est important d'introduire le concept d'arrays de booléens. Jusqu'à maintenant nous avions définis uniquement des arrays avec des types numériques int ou float. Il est tout à fait possible de définir des arrays de booléens. La fonction np.full() vue ci-dessus nous permet d'en construire facilement :
>>> np.full((2, 2), True)
array([[ True, True],
[ True, True]])
>>> np.full((2, 2), False)
array([[False, False],
[False, False]])
Très bien, mais au premier abord nous n'en voyons pas forcément l'utilité... Mais qu'en est-il lorsqu'on utilise les opérateurs de comparaison avec un array ? Et bien cela renvoie un array de booléens !
>>> a = np.reshape(np.arange(1, 10), (3, 3))
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> a > 5
array([[False, False, False],
[False, False, True],
[ True, True, True]])
>>> a == 2
array([[False, True, False],
[False, False, False],
[False, False, False]])
Tous les éléments de l'array satisfaisant la condition seront à True, les autres à False. Encore plus fort, il est possible de combiner plusieurs conditions avec les opérateurs logiques & et | (respectivement ET et OU) :
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> (a > 3) & (a % 2 == 0)
array([[False, False, False],
[ True, False, True],
[False, True, False]])
>>> (a > 3) | (a % 2 == 0)
array([[False, True, False],
[ True, True, True],
[ True, True, True]])
Nous pouvons effectuer deux remarques :
- Les opérateurs logiques
&et|s'appliquent sur les arrays et sont différents des opérateurs logiquesandetorqui eux s'appliquent sur les booléens (TrueouFalse) ; - Il est conseillé de mettre des parenthèses pour chaque condition afin d'éviter les ambiguïtés.
Maintenant que les arrays de booléens ont été introduits, nous pouvons définir les masques booléens :
Les masques booléens sont des arrays de booléens qui sont utilisés en tant qu'« indice » d'un array initial. Cela permet de récupérer / modifier une partie de l'array initial.
Concrètement, il suffira d'utiliser un array et un opérateur de comparaison entre les crochets qui étaient dédiés à l'indiçage :
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> a[a > 5]
array([6, 7, 8, 9])
>>> a[a == 2]
array([2])
>>> a[a != 0]
array([1, 2, 3, 4, 5, 6, 7, 8, 9])
On voit que l'on récupère seulement les éléments de l'array a qui sastisfont la sélection ! Toutefois, il est important de constater que l'array renvoyé perd la dimensionnalité de l'array a initial, il s'agit systématiquement d'un array 1D.
La grande puissance de ce mécanisme est que l'on peut utiliser les masques booléens pour modifier les éléments que l'on sélectionne :
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> a[a > 5]
array([6, 7, 8, 9])
>>> a[a > 5] = -1
>>> a
array([[ 1, 2, 3],
[ 4, 5, -1],
[-1, -1, -1]])
On peut bien sûr combiner plusieurs conditions avec les opérateurs logiques :
>>> a
array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
>>> a[(a > 3) | (a % 2 == 0)] = 0
>>> a
array([[1, 0, 3],
[0, 0, 0],
[0, 0, 0]])
Ce mécanisme de sélection avec des masques booléens se révèle très puissant pour manipuler des grandes quantités de données. On verra qu'il peut être également utilisé avec les dataframes du module pandas.
Les masques booléens ne doivent pas être confondus avec les masked arrays qui sont des arrays dans lesquels on peut trouver des valeurs manquantes ou invalides.
Une application possible des masques est de « binariser » une matrice de nombre :
>>> import random as rd
>>> import numpy as np
>>> a = np.resize([rd.random() for i in range(16)], (4,
4))
>>> a
array([[0.58704728, 0.50212977, 0.70652863, 0.24158108],
[0.93102132, 0.41864373, 0.45807961, 0.98288744],
[0.48198211, 0.16877376, 0.14431518, 0.74784176],
[0.92913469, 0.08383269, 0.10670144, 0.14554345]])
>>> seuil = 0.3
>>> a[a < seuil] = 0
>>> a[a > seuil] = 1
>>> a
array([[1., 1., 1., 0.],
[1., 1., 1., 1.],
[1., 0., 0., 1.],
[1., 0., 0., 0.]])
En deux lignes de code cela est fait alors qu'il aurait fallu faire des boucles avec les listes classiques !
17.1.11 Quelques conseils¶
Nous venons de voir une petite partie du module NumPy mais vous avez pu en constater son extraordinaire puissance. On pourrait au premier abord être tenté d'abandonner les listes, toutefois elles gardent toute leur importance. Alors quand utiliser les listes ou quand utiliser les arrays NumPy ? Voici une liste non exhaustive d'éléments qui peuvent guider votre choix :
Utilisez NumPy pour :
- les opérations vectorielles (éléments par éléments) ;
- lorsque vous souhaitez manipuler des objets mathématiques (vecteurs, matrices, etc.) et les outils associés (algèbre linéaire) ;
- tout ce qui est numérique de manière générale.
Utilisez les listes :
- lorsque vous avez besoin d'un container pour accumuler des valeurs (fussent-elles des sous-listes), surtout lorsqu'elles ne sont pas homogènes (i.e. du même type) ;
- lorsque vous souhaitez accumuler des valeurs au fur et à mesure des itérations d'une boucle : la méthode
.append()des listes est bien plus efficace que faire grandir un array ligne par ligne (i.e. en ajoutant une ligne avecnp.concatenate()à chaque tour de boucle) ; lorsqu'on ne peut pas utiliser les fonctions de lecture de fichier de NumPy pour quelque raison que ce soit, il est tout à fait classique de faire grandir une liste au fur et à mesure de la lecture du fichier puis de la convertir à la fin en array. De manière générale, utiliseznp.concatenate()seulement pour concaténer des gros arrays, pas pour ajouter juste une ligne.
Enfin, comme nous vous le conseillons depuis le début, soignez bien votre documentation (docstrings) ou vos commentaires lorsque vous utilisez des arrays NumPy. NumPy permettant de réaliser des opérations vectorielles de manière très compacte, il est essentiel de se mettre à la place du lecteur et de bien indiquer ce que contient chaque array et sa dimensionnalité (1D, 2D, etc.).
Le module NumPy est la brique de base pour tout ce qui est numérique. Associé aux modules SciPy et matplotlib (et aussi aux notebooks Jupyter, voir le chapitre suivant), il permet de faire de l'analyse scientifique. On verra un peu plus loin dans ce chapitre que la puissance de NumPy est également utilisée par le module pandas et ses fameux dataframes pour faire de l'analyse de données.
- Le livre de Nicolas Rougier From Python to Numpy est une très bonne ressource pour explorer plus en détails les possibilités de NumPy. L'auteur y aborde notamment la puissante notion de « masque ».
- Les tutoriels proposés par l'équipe de développement de NumPy sont également un excellent moyen de poursuivre votre découverte de cette bibliothèque incontournable en sciences.
17.2 Module Biopython¶
Le module Biopython propose des fonctionnalités très utiles en bioinformatique. Le tutoriel est particulièrement bien fait, n'hésitez pas à le consulter.
17.2.1 Manipulation de données¶
Voici quelques exemples de manipulation de données avec Biopython.
Définition d'une séquence :
>>> import Bio
>>> from Bio.Seq import Seq
>>> ADN = Seq("ATATCGGCTATAGCATGC")
>>> ADN
Seq('ATATCGGCTATAGCATGC')
Ligne 1. Le module Biopython s'appelle Bio.
Obtention de la séquence complémentaire et complémentaire inverse :
>>> ADN.complement()
Seq('TATAGCCGATATCGTACG')
>>> ADN.reverse_complement()
Seq('GCATGCTATAGCCGATAT')
Traduction en séquence protéique :
Dans l'annexe A Quelques formats de données rencontrés en biologie, vous trouverez de nombreux exemples d'utilisation de Biopython pour manipuler des données aux formats FASTA, GenBank et PDB.17.2.2 Interrogation de la base de données PubMed¶
Le sous-module Entrez de Biopython permet d’utiliser les ressources du NCBI et notamment d'interroger le site PubMed.
Nous allons par exemple utiliser PubMed pour chercher des articles scientifiques relatifs à la transferrine (transferrin en anglais):
>>> from Bio import Entrez
>>> Entrez.email = "votremail@provider.fr"
>>> req_esearch = Entrez.esearch(db="pubmed", term="transferrin")
>>> res_esearch = Entrez.read(req_esearch)
Ligne 1. On charge directement le sous-module Entrez.
Ligne 2. Lors d'une requête sur le site du NCBI, il est important de définir correctement
la variable Entrez.email qui sera transmise au NCBI lors de la requête et qui
pourra être utilisée pour vous contacter en cas de difficulté avec le serveur.
Ligne 3. On lance la requête (transferrin) sur le moteur de recherche pubmed. La requête est stockée dans la variable req_esearch.
Ligne 4. Le résultat est lu et stocké dans la variable res_esearch.
Sans être un vrai dictionnaire, la variable res_esearch en a cependant plusieurs propriétés.
Voici ses clés :
>>> res_esearch.keys()
dict_keys(['Count', 'RetMax', 'RetStart', 'IdList', 'TranslationSet',
'TranslationStack', 'QueryTranslation'])
La valeur associée à la clé IdList est une liste qui contient les identifiants
(PMID) des articles scientifiques associés à la requête (ici transferrin) :
>>> res_esearch["IdList"]
['30411489', '30409795', '30405884', '30405827', '30402883', '30401570',
'30399508', '30397276', '30395963', '30394734', '30394728', '30394123',
'30393423', '30392910', '30392664', '30391706', '30391651', '30391537',
'30391296', '30390672']
>>> len(res_esearch["IdList"])
20
Cette liste ne contient les identifiants que de 20 publications alors que si nous faisons cette même requête directement sur le site de PubMed depuis un navigateur web, nous obtenons plus de 33900 résultats.
En réalité, le nombre exact de publications est connu :
Pour ne pas saturer les serveurs du NCBI, seulement 20 PMID sont renvoyés par défaut.
Mais vous pouvez augmenter cette limite en utilisant le paramètre retmax dans la fonction Entrez.esearch().
Nous pouvons maintenant récupérer des informations sur une publication précise en connaissant son PMID. Par exemple, l'article avec le PMID 22294463 dont un aperçu est sur la figure 2.

Nous allons pour cela utiliser la fonction Entrez.esummary()
>>> req_esummary = Entrez.esummary(db="pubmed", id="22294463")
>>> res_esummary = Entrez.read(req_esummary)
La variable res_esummary n'est pas réellement une liste mais en a plusieurs propriétés.
Cette pseudo-liste n'a qu'un seul élément, qui est lui-même un pseudo-dictionnaire dont voici les clés :
>>> res_esummary[0].keys()
dict_keys(['Item', 'Id', 'PubDate', 'EPubDate', 'Source', 'AuthorList',
'LastAuthor', 'Title', 'Volume', 'Issue', 'Pages', 'LangList',
'NlmUniqueID', 'ISSN', 'ESSN', 'PubTypeList', 'RecordStatus', 'PubStatus',
'ArticleIds', 'DOI', 'History', 'References', 'HasAbstract', 'PmcRefCount',
'FullJournalName', 'ELocationID', 'SO'])
Nous pouvons alors facilement obtenir le titre, le DOI et la date de publication
(PubDate) de cet article, ainsi que le journal (Source) dans lequel il a été
publié :
>>> res_esummary[0]["Title"]
'Known and potential roles of transferrin in iron biology.'
>>> res_esummary[0]["DOI"]
'10.1007/s10534-012-9520-3'
>>> res_esummary[0]["PubDate"]
'2012 Aug'
>>> res_esummary[0]["Source"]
'Biometals'
Enfin, pour récupérer le résumé de la publication précédente, nous allons
utiliser la fonction Entrez.efetch() :
>>> req_efetch = Entrez.efetch(db="pubmed", id="22294463", rettype="txt")
>>> res_efetch = Entrez.read(req_efetch)
La variable res_efetch est un pseudo-dictionnaire qui contient une pseudo-liste,
qui contient un pseudo-dictionnaire, qui contient... Oui, c'est compliqué !
Pour faire court, le résumé peut s'obtenir avec l'instruction :
>>> res_efetch['PubmedArticle'][0]['MedlineCitation']['Article'] \
... ['Abstract']['AbstractText'][0]
'Transferrin is an abundant serum metal-binding protein best known
for its role in iron delivery. The human disease congenital atransf
errinemia and animal models of this disease highlight the essential
role of transferrin in erythropoiesis and iron metabolism. Patient
s and mice deficient in transferrin exhibit anemia and a paradoxica
l iron overload attributed to deficiency in hepcidin, a peptide hor
mone synthesized largely by the liver that inhibits dietary iron ab
sorption and macrophage iron efflux. Studies of inherited human dis
ease and model organisms indicate that transferrin is an essential
regulator of hepcidin expression. In this paper, we review current
literature on transferrin deficiency and present our recent finding
s, including potential overlaps between transferrin, iron and manga
nese in the regulation of hepcidin expression.'
Ce qui est bien le résumé que nous obtenons sur la figure 2.
17.3 Module matplotlib¶
Le module matplotlib permet de générer des graphiques depuis Python. Il est l'outil complémentaire de NumPy, scipy ou pandas (que l'on verra juste après) lorsqu'on veut faire de l'analyse de données.
17.3.1 Représentation sous forme de points¶
Dans cet exemple, nous considérons l'évolution de la concentration d'un produit dans le sang (exprimé en mg/L) en fonction du temps (exprimé en heure).
Nous avons les résultats suivants :
| Temps (h) | Concentration (mg/L) |
|---|---|
| 1 | 3.5 |
| 2 | 5.8 |
| 3 | 9.1 |
| 4 | 11.8 |
| 6 | 17.5 |
| 7 | 21.3 |
| 9 | 26.8 |
Nous allons maintenant représenter l'évolution de la concentration en fonction du temps :
import matplotlib.pyplot as plt
temps = [1, 2, 3, 4, 6, 7, 9]
concentration = [5.5, 7.2, 11.8, 13.6, 19.1, 21.7, 29.4]
plt.scatter(temps, concentration, marker="o", color="blue")
plt.xlabel("Temps (h)")
plt.ylabel("Concentration (mg/L)")
plt.title("Concentration de produit en fonction du temps")
plt.show()
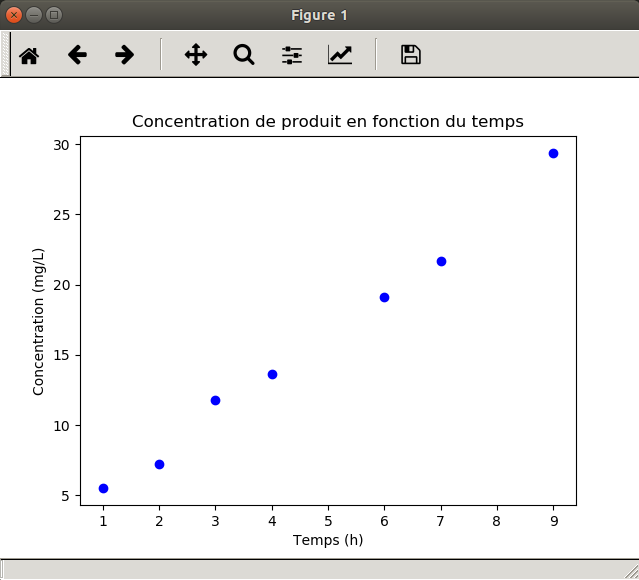
Revenons maintenant sur le code.
Ligne 1. Tout d'abord, on importe le sous-module pyplot du module matplotlib et on lui donne l'alias plt pour l'utiliser plus rapidement ensuite (cet alias est standard, utilisez-la systématiquement).
Lignes 3 et 4. On définit les variables temps et concentration comme des listes. Les deux listes doivent avoir la même longueur (7 éléments dans le cas présent).
Ligne 5. La fonction scatter() permet de représenter des points sous forme de nuage de points. Les deux premiers arguments correspondent aux valeurs en abscisse et en ordonnée des points, fournis sous forme de listes. Des arguments facultatifs sont ensuite précisés comme le symbole (marker) et la couleur (color).
Lignes 6 et 7. Les fonctions xlabel() et ylabel() sont utilisées pour donner un nom aux axes.
Ligne 8. La fonction title() définit le titre du graphique.
Ligne 9. Enfin, la fonction show() affiche le graphique généré à l'écran.
17.3.2 Représentation sous forme de courbe¶
On sait par ailleurs que l'évolution de la concentration du produit en fonction du temps peut-être modélisée par la fonction \(f(x) = 2 + 3 \times x\). Représentons ce modèle avec les points expérimentaux et sauvegardons le graphique obtenu sous forme d'une image :
import numpy as np
import matplotlib.pyplot as plt
temps = [1, 2, 3, 4, 6, 7, 9]
concentration = [5.5, 7.2, 11.8, 13.6, 19.1, 21.7, 29.4]
plt.scatter(temps, concentration, marker="o", color = "blue")
plt.xlabel("Temps (h)")
plt.ylabel("Concentration (mg/L)")
plt.title("Concentration de produit en fonction du temps")
x = np.linspace(min(temps), max(temps), 50)
y = 2 + 3 * x
plt.plot(x, y, color='green', ls="--")
plt.grid()
plt.savefig("concentration_vs_temps.png", bbox_inches='tight', dpi=200)
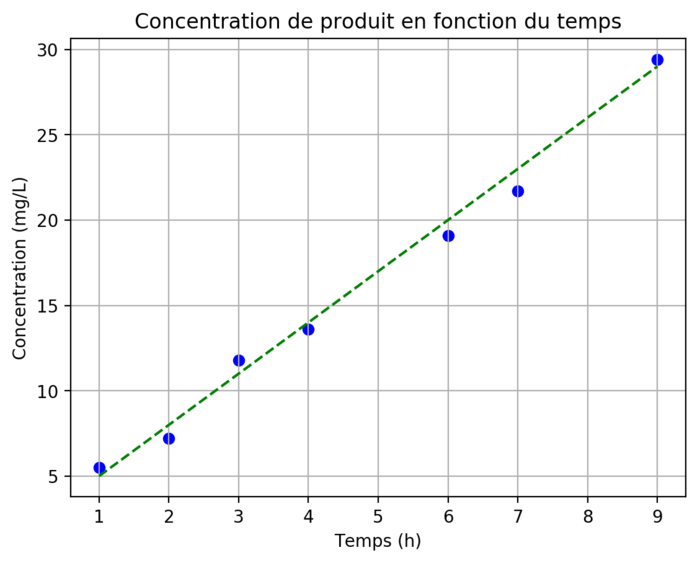 { #fig:conc-vs-tps width=70% }
{ #fig:conc-vs-tps width=70% }
Les étapes supplémentaires par rapport au graphique précédent (figure 3) sont :
Ligne 1. On charge le module numpy sous le nom np.
Ligne 10. On crée la variable x avec la fonction linspace() du module NumPy qui renvoie une liste de valeurs régulièrement espacées entre deux bornes, ici entre le minimum (min(temps)) et le maximum (max(temps)) de la variable temps. Dans notre exemple, nous générons une liste de 50 valeurs. La variable x ainsi créée est du type array.
Ligne 11. On construit ensuite la variable y à partir de la formule modélisant l'évolution de la concentration en fonction du temps. Cette manipulation n'est possible que parce que x est du type array. Cela ne fonctionnerait pas avec une liste classique.
Ligne 12. La fonction plot() permet de construire une courbe à partir des coordonnées en abscisse et en ordonnées des points à représenter. On indique ensuite des arguments facultatifs comme le style de la ligne (ls) et sa couleur (color).
Ligne 13. La fonction grid() affiche une grille.
Ligne 14. Enfin, la fonction savefig() enregistre le graphique produit sous la forme d'une image au format png. Des arguments par mot-clé définissent la manière de générer les marges autour du graphique (bbox_inches) et la résolution de l'image (dpi).
17.3.3 Représentation sous forme de diagramme en bâtons¶
On souhaite maintenant représenter graphiquement la distribution des différentes bases dans une séquence d'ADN.
import numpy as np
import matplotlib.pyplot as plt
sequence = "ACGATCATAGCGAGCTACGTAGAA"
bases = ["A", "C", "G", "T"]
distribution = []
for base in bases:
distribution.append(sequence.count(base))
x = np.arange(len(bases))
plt.bar(x, distribution)
plt.xticks(x, bases)
plt.xlabel("Bases")
plt.ylabel("Nombre")
plt.title(f"Distribution des bases\n dans la séquence {sequence}")
plt.savefig("distribution_bases.png", bbox_inches="tight", dpi=200)
On obtient alors le graphique de la figure 5.
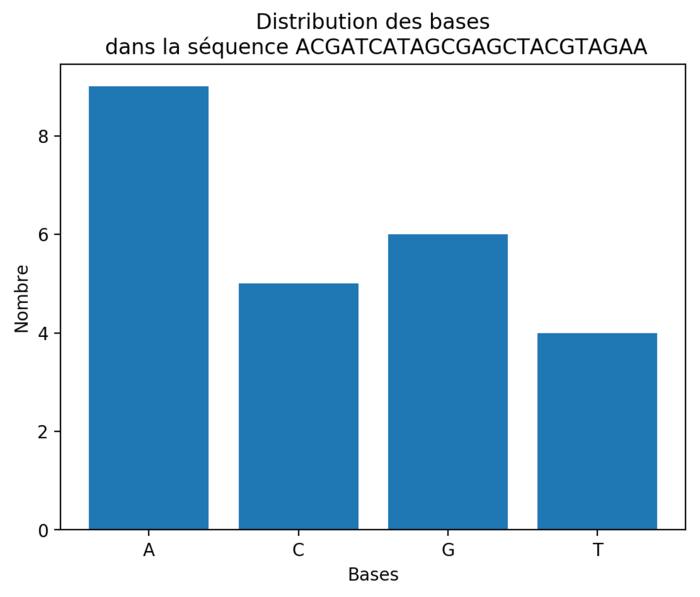
Prenons le temps d'examiner les différentes étapes du script précédent :
Lignes 4 à 6. On définit les variables sequence, bases et distribution qu'on utilise ensuite.
Lignes 7 et 8. On calcule la distribution des différentes bases dans la séquence. On utilise pour cela la méthode count() qui renvoie le nombre de fois qu'une chaîne de caractères (les différentes bases) se trouve dans une autre (la séquence).
Ligne 10. On définit la position en abscisse des barres. Dans cet exemple, la variable x vaut array([0, 1, 2, 3]).
Ligne 11. La fonction bar() construit le diagramme en bâtons. Elle prend en argument la position des barres (x) et leurs hauteurs (distribution).
Ligne 12. La fonction xtics() redéfinit les étiquettes (c'est-à-dire le nom des bases) sur l'axe des abscisses.
Lignes 13 à 15. On définit les légendes des axes et le titre du graphique. On insère un retour à la ligne \n dans le titre pour qu'il soit réparti sur deux lignes.
Ligne 16. Enfin, on enregistre le graphique généré au format png.
On espère que ces courts exemples vous auront convaincu de l'utilité du module matplotlib. Sachez qu'il peut faire bien plus, par exemple générer des histogrammes ou toutes sortes de graphiques utiles en analyse de données.
- Le site de matplotlib fournit de nombreux exemples détaillés, n'hésitez pas à le consulter.
- Le site Python Graph Gallery propose aussi des exemples de code pour différents types de graphiques.
- Enfin, des cheat sheets de matplotlib sont aussi disponibles.
17.4 Module pandas¶
Le module pandas a été conçu pour l'analyse de données. Il est particulièrement puissant pour manipuler des données structurées sous forme de tableau.
Pour charger pandas dans la mémoire de Python, on utilise la commande import habituelle :
Pandas est souvent chargé avec un nom raccourci, comme pour NumPy et matplotlib :
17.4.1 Series¶
Le premier type de données apporté par pandas est la series, qui correspond à un vecteur à une dimension.
>>> s = pd.Series([10, 20, 30, 40], index = ['a', 'b', 'c', 'd'])
>>> s
a 10
b 20
c 30
d 40
dtype: int64
Avec pandas, chaque élément de la série de données possède une étiquette qui permet d'appeler les éléments.
Ainsi, pour appeler le premier élément de la série, on peut utiliser son index,
comme pour une liste (0 pour le premier élément) ou son étiquette (ici, "a") :
Bien sûr, on peut extraire plusieurs éléments, par leurs indices ou leurs étiquettes :
Les étiquettes permettent de modifier et d'ajouter des éléments :
Enfin, on peut filtrer une partie de la series :
et même combiner plusieurs critères de sélection :
17.4.2 Dataframes¶
Un autre type d'objet particulièrement intéressant introduit par pandas sont les dataframes. Ceux-ci correspondent à des tableaux à deux dimensions avec des étiquettes pour nommer les lignes et les colonnes.
Si vous êtes familier avec le langage de programmation et d'analyse statistique R, les dataframes de pandas se rapprochent de ceux trouvés dans R.
Voici comment créer un dataframe avec pandas à partir de données fournies comme liste de lignes :
>>> df = pd.DataFrame(columns=["a", "b", "c", "d"],
... index=["chat", "singe", "souris"],
... data=[np.arange(10, 14),
... np.arange(20, 24),
... np.arange(30, 34)])
>>> df
a b c d
chat 10 11 12 13
singe 20 21 22 23
souris 30 31 32 33
Ligne 1. Le dataframe est créé avec la fonction DataFrame() à laquelle
on fournit plusieurs arguments.
L'argument columns indique le nom des colonnes, sous forme d'une liste.
Ligne 2. L'argument index définit le nom des lignes, sous forme de liste.
Lignes 3-5. L'argument data fournit le contenu du dataframe, sous la forme
d'une liste de valeurs correspondantes à des lignes. Ainsi np.arange(10, 14)
qui est équivalent à [10, 11, 12, 13] correspond à la première ligne du dataframe.
Notez ici qu'il faut avoir préalablement importer numpy avec l'instruction import numpy as np.
Le même dataframe peut aussi être créé à partir des valeurs fournies en colonnes sous la forme d'un dictionnaire :
>>> data = {"a": np.arange(10, 40, 10),
... "b": np.arange(11, 40, 10),
... "c": np.arange(12, 40, 10),
... "d": np.arange(13, 40, 10)}
>>> df = pd.DataFrame(data)
>>> df.index = ["chat", "singe", "souris"]
>>> df
a b c d
chat 10 11 12 13
singe 20 21 22 23
souris 30 31 32 33
Lignes 1-4. Le dictionnaire data contient les données en colonnes.
La clé associée à chaque colonne est le nom de la colonne.
Ligne 5. Le dataframe est créé avec la fonction pd.DataFrame()
à laquelle on passe data en argument.
Ligne 6. On peut définir les étiquettes des lignes de n'importe quel dataframe
avec l'attribut df.index.
17.4.3 Quelques propriétés¶
Les dimensions d'un dataframe sont données par l'attribut .shape :
Ici, le dataframe df a 3 lignes et 4 colonnes.
L'attribut .columns renvoie le nom des colonnes et permet aussi de renommer
les colonnes d'un dataframe :
>>> df.columns
Index(['a', 'b', 'c', 'd'], dtype='object')
>>> df.columns = ["Paris", "Lyon", "Nantes", "Pau"]
>>> df
Paris Lyon Nantes Pau
chat 10 11 12 13
singe 20 21 22 23
souris 30 31 32 33
La méthode .head(n) renvoie les n premières lignes du dataframe
(par défaut, n vaut 5) :
17.4.4 Sélection¶
Les mécanismes de sélection fournis avec pandas sont très puissants. En voici un rapide aperçu :
Sélection de colonnes¶
On peut sélectionner une colonne par son étiquette :
ou plusieurs colonnes en même temps :
Pour la sélection de plusieurs colonnes, les étiquettes d'intérêt sont rassemblées dans une liste.Sélection de lignes¶
Pour sélectionner une ligne, il faut utiliser l'instruction .loc()
et l'étiquette de la ligne :
Ici aussi, on peut sélectionner plusieurs lignes :
Enfin, on peut aussi sélectionner des lignes avec l'instruction .iloc
et l'indice de la ligne (la première ligne ayant l'indice 0) :
On peut également utiliser les tranches (comme pour les listes) :
Sélection sur les lignes et les colonnes¶
On peut bien sûr combiner les deux types de sélection (en ligne et en colonne):
>>> df.loc["souris", "Pau"]
33
>>> df.loc[["singe", "souris"], ['Nantes', 'Lyon']]
Nantes Lyon
singe 22 21
souris 32 31
Notez qu'à partir du moment où on souhaite effectuer une sélection sur des lignes,
il faut utiliser loc (ou iloc si on utilise les indices).
Sélection par condition¶
Remémorons-nous d'abord le contenu du dataframe df :
Sélectionnons maintenant toutes les lignes pour lesquelles les effectifs à Pau sont supérieurs à 15 :
De cette sélection, on ne souhaite garder que les valeurs pour Lyon :
On peut aussi combiner plusieurs conditions avec & pour l'opérateur et :
et | pour l'opérateur ou :
>>> df[ (df["Pau"]>15) | (df["Lyon"]>25) ]
Paris Lyon Nantes Pau
singe 20 21 22 23
souris 30 31 32 33
17.4.5 Combinaison de dataframes¶
En biologie, on a souvent besoin de combiner deux tableaux de chiffres à partir d'une colonne commune.
Par exemple, si on considère les deux dataframes suivants :
>>> data1 = {"Lyon": [10, 23, 17], "Paris": [3, 15, 20]}
>>> df1 = pd.DataFrame.from_dict(data1)
>>> df1.index = ["chat", "singe", "souris"]
>>> df1
Lyon Paris
chat 10 3
singe 23 15
souris 17 20
et
>>> data2 = {"Nantes": [3, 9, 14], "Strasbourg": [5, 10, 8]}
>>> df2 = pd.DataFrame.from_dict(data2)
>>> df2.index = ["chat", "souris", "lapin"]
>>> df2
Nantes Strasbourg
chat 3 5
souris 9 10
lapin 14 8
On souhaite combiner ces deux dataframes, c'est-à-dire connaître pour les 4 villes (Lyon, Paris, Nantes et Strasbourg) le nombre d'animaux. On remarque d'ores et déjà qu'il y a des singes à Lyon et Paris mais pas de lapin et qu'il y a des lapins à Nantes et Strasbourg mais pas de singe. Nous allons voir comment gérer cette situation.
pandas propose pour cela la fonction concat() qui prend comme argument une liste de dataframes :
>>> pd.concat([df1, df2])
Lyon Nantes Paris Strasbourg
chat 10.0 NaN 3.0 NaN
singe 23.0 NaN 15.0 NaN
souris 17.0 NaN 20.0 NaN
chat NaN 3.0 NaN 5.0
souris NaN 9.0 NaN 10.0
lapin NaN 14.0 NaN 8.0
Ici, NaN indique des valeurs manquantes. Mais le résultat obtenu n'est pas celui que nous attendions puisque les lignes de deux dataframes ont été recopiées.
L'argument supplémentaire axis=1 produit le résultat attendu :
>>> pd.concat([df1, df2], axis=1)
Lyon Paris Nantes Strasbourg
chat 10.0 3.0 3.0 5.0
lapin NaN NaN 14.0 8.0
singe 23.0 15.0 NaN NaN
souris 17.0 20.0 9.0 10.0
Par défaut, pandas va conserver le plus de lignes possible. Si on ne souhaite conserver que les lignes communes aux deux dataframes, il faut ajouter l'argument join="inner" :
>>> pd.concat([df1, df2], axis=1, join="inner")
Lyon Paris Nantes Strasbourg
chat 10 3 3 5
souris 17 20 9 10
Un autre comportement par défaut de concat() est que cette fonction va combiner les dataframes en se basant sur leurs index. Il est néanmoins possible de préciser, pour chaque dataframe, le nom de la colonne qui sera utilisée comme référence avec l'argument join_axes.
17.5 Un exemple plus complet¶
Pour illustrer les possibilités de pandas, voici un exemple plus complet.
Le fichier transferrin_report.csv que vous pouvez télécharger
ici
contient une liste de structures de la transferrine.
Cette protéine est responsable du transport du fer dans l'organisme.
Si vous n'êtes pas familier avec le format de fichier .csv, nous vous conseillons
de consulter l'annexe A Quelques formats de données rencontrés en biologie.
Voyons maintenant comment explorer les données contenues dans ce fichier avec pandas.
17.5.1 Prise de contact avec le jeu de données¶
Une fonctionnalité très intéressante de pandas est d'ouvrir très facilement
un fichier au format .csv :
Le contenu est chargé sous la forme d'un dataframe dans la variable df.
Le fichier contient 41 lignes de données plus une ligne d'en-tête. Cette dernière est automatiquement utilisée par pandas pour nommer les différentes colonnes. Voici un aperçu des premières lignes :
>>> df.head()
PDB ID Source Deposit Date Length MW
0 1A8E Homo sapiens 1998-03-24 329 36408.4
1 1A8F Homo sapiens 1998-03-25 329 36408.4
2 1AIV Gallus gallus 1997-04-28 686 75929.0
3 1AOV Anas platyrhynchos 1996-12-11 686 75731.8
4 1B3E Homo sapiens 1998-12-09 330 36505.5
Nous avons 5 colonnes de données :
- l'identifiant de la structure (
PDB ID) ; - l'organisme d'où provient cette protéine (
Source) ; - la date à laquelle cette structure a été déposée dans la Protein Data Bank (
Deposit Date) ; - le nombre d'acides aminés qui constituent la protéine (
Length) ; - et la masse molaire de la protéine (
MW).
La colonne d'entiers tout à gauche est un index automatiquement créé par pandas.
Nous pouvons demander à pandas d'utiliser une colonne particulière comme index.
La colonne PDB ID s'y prête très bien car cette colonne ne contient que
des identifiants uniques :
>>> df = pd.read_csv("transferrin_report.csv", index_col="PDB ID")
>>> df.head()
Source Deposit Date Length MW
PDB ID
1A8E Homo sapiens 1998-03-24 329 36408.4
1A8F Homo sapiens 1998-03-25 329 36408.4
1AIV Gallus gallus 1997-04-28 686 75929.0
1AOV Anas platyrhynchos 1996-12-11 686 75731.8
1B3E Homo sapiens 1998-12-09 330 36505.5
Avant d'analyser un jeu de données, il est intéressant de l'explorer un peu. Par exemple, connaître ses dimensions :
Notre jeu de données contient donc 41 lignes et 4 colonnes. En effet,
la colonne PDB ID est maintenant utilisée comme index et n'est donc plus
prise en compte.
Il est aussi intéressant de savoir de quel type de données est constituée chaque colonne :
Les colonnes Length et MW contiennent des valeurs numériques,
respectivement des entiers (int64) et des floats (float64).
Le type object est un type par défaut.
La méthode .info() permet d'aller un peu plus loin dans l'exploration du jeu de données
en combinant les informations produites par les propriétés .shape et .dtypes :
>>> df.info()
<class 'pandas.core.frame.DataFrame'>
Index: 41 entries, 1A8E to 6CTC
Data columns (total 4 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Source 41 non-null object
1 Deposit Date 41 non-null object
2 Length 41 non-null int64
3 MW 41 non-null float64
dtypes: float64(1), int64(1), object(2)
memory usage: 1.6+ KB
Avec l'argument memory_usage="deep", cette méthode permet surtout de connaitre avec précision
la taille de l'espace mémoire occupé par le dataframe :
>>> df.info(memory_usage="deep")
<class 'pandas.core.frame.DataFrame'>
Index: 41 entries, 1A8E to 6CTC
Data columns (total 4 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Source 41 non-null object
1 Deposit Date 41 non-null object
2 Length 41 non-null int64
3 MW 41 non-null float64
dtypes: float64(1), int64(1), object(2)
memory usage: 8.6 KB
Ici, le dataframe occupe 8,6 ko en mémoire.
17.5.2 Conversion en date¶
Le type object correspond la plupart du temps à des chaînes de caractères.
C'est tout à fait légitime pour la colonne Source. Mais on sait par contre
que la colonne Deposit Date est une date sous la forme
année-mois-jour.
Si le format de date utilisé est homogène sur tout le jeu de données et non ambigu,
pandas va se débrouiller pour trouver automatiquement le format de date utilisé.
On peut alors explicitement demander à pandas de considérer la colonne
Deposit Date comme une date :
L'affichage des données n'est pas modifié :
>>> df.head()
Source Deposit Date Length MW
PDB ID
1A8E Homo sapiens 1998-03-24 329 36408.4
1A8F Homo sapiens 1998-03-25 329 36408.4
1AIV Gallus gallus 1997-04-28 686 75929.0
1AOV Anas platyrhynchos 1996-12-11 686 75731.8
1B3E Homo sapiens 1998-12-09 330 36505.5
Mais le type de données de la colonne Deposit Date est maintenant une date (datetime64[ns]) :
17.5.3 Statistiques descriptives et table de comptage¶
Pour les colonnes qui contiennent des données numériques, on peut obtenir
rapidement quelques statistiques descriptives avec la méthode .describe() :
>>> df.describe()
Length MW
count 41.000000 41.000000
mean 477.341463 52816.090244
std 175.710217 19486.594012
min 304.000000 33548.100000
25% 331.000000 36542.300000
50% 337.000000 37229.300000
75% 679.000000 75298.500000
max 696.000000 77067.900000
On apprend ainsi que la masse moléculaire (colonne MW)
a une valeur moyenne de 52816.090244 avec un écart-type de 19486.594012 et que
la plus petite valeur est 33548.100000 et la plus grande 77067.900000. Pratique !
La colonne Source contient des chaînes de caractères, on peut rapidement
déterminer le nombre de protéines pour chaque organisme :
>>> df["Source"].value_counts()
Homo sapiens 26
Gallus gallus 10
Anas platyrhynchos 2
Oryctolagus cuniculus 2
Sus scrofa 1
Name: Source, dtype: int64
Ainsi, 26 protéines sont d'origine humaine (Homo sapiens) et 10 proviennent
de la poule (Gallus gallus).
17.5.4 Statistiques par groupe¶
On peut aussi déterminer, pour chaque organisme, la taille et la masse moléculaire moyennes des transferrines :
>>> df.groupby(["Source"]).mean()
Length MW
Source
Anas platyrhynchos 686.000000 75731.800000
Gallus gallus 509.300000 56324.080000
Homo sapiens 439.615385 48663.392308
Oryctolagus cuniculus 490.000000 54219.600000
Sus scrofa 696.000000 77067.900000
La méthode .groupby() rassemble d'abord les données suivant la colonne
Source puis la méthode .mean() calcule la moyenne pour chaque groupe.
Si on souhaite obtenir deux statistiques (par exemple la valeur minimale et maximale)
en une seule fois, il convient alors d'utiliser la méthode .pivot_table()
plus complexe mais aussi beaucoup plus puissante :
>>> df.pivot_table(index="Source", values=["Length", "MW"], aggfunc=[min, max])
min max
Length MW Length MW
Source
Anas platyrhynchos 686 75731.8 686 75731.8
Gallus gallus 328 36105.8 686 75957.1
Homo sapiens 327 36214.2 691 76250.2
Oryctolagus cuniculus 304 33548.1 676 74891.1
Sus scrofa 696 77067.9 696 77067.9
L'argument index précise la colonne dont on agrège les données.
L'argument values indique sur quelles colonnes les statistiques sont calculées.
Enfin, aggfunc liste les statistiques calculées, ici la valeur minimale et maximale.
Notez que les valeurs renvoyées sont d'abord les valeurs minimales pour Length
et MW puis les valeurs maximales pour Length et MW.
17.5.5 Analyse de données numériques¶
On peut, sans trop de risque, émettre l'hypothèse que plus il y a d'acides aminés dans la protéine, plus sa masse moléculaire va être élevée.
Pour vérifier cela graphiquement, on représente la masse moléculaire de la protéine en fonction de sa taille (c'est-à-dire du nombre d'acides aminés).
>>> import matplotlib.pyplot as plt
>>> plt.scatter(df["Length"], df["MW"])
<matplotlib.collections.PathCollection object at 0x7f62c2501780>
>>> plt.xlabel("Taille (nombre d'acides aminés)")
Text(0.5, 0, "Taille (nombre d'acides aminés)")
>>> plt.ylabel("Masse moléculaire (Dalton)")
Text(0, 0.5, 'Masse moléculaire (Dalton)')
>>> plt.savefig("transferrine1.png")
On obtient un graphique similaire à celui de la figure 6 (A) avec deux groupes de points distincts (car certaines structures sont incomplètes).
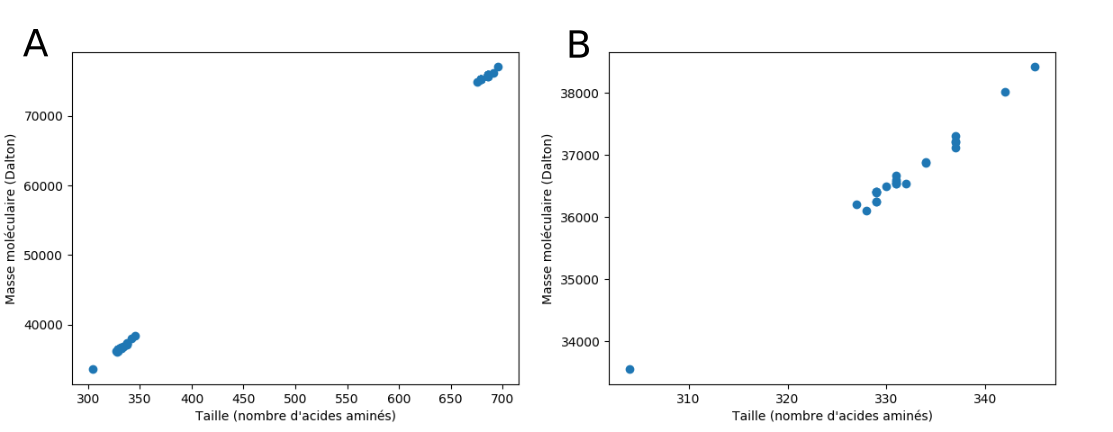
On peut zoomer sur le groupe de points le plus à gauche en ne sélectionnant que les protéines constituées de moins de 400 résidus :
Puis en créant un deuxième graphique :>>> plt.clf()
>>> plt.scatter(dfz["Length"], dfz["MW"])
<matplotlib.collections.PathCollection object at 0x7f85bb4852e8>
>>> plt.xlabel("Taille (nombre d'acides aminés)")
Text(0.5, 0, "Taille (nombre d'acides aminés)")
>>> plt.ylabel("Masse moléculaire (Dalton)")
Text(0, 0.5, 'Masse moléculaire (Dalton)')
>>> plt.savefig("transferrine2.png")
Ligne 1. L'instruction plt.clf() efface le graphe précédent mais conserve les
noms des axes des abscisses et des ordonnées.
Le graphique 6 (B) obtenu met en évidence une relation linéaire entre le nombre de résidus d'une protéine et sa masse moléculaire.
En réalisant une régression linéaire, on détermine les paramètres de la droite qui passent le plus proche possible des points du graphique.
>>> from scipy.stats import linregress
>>> lr = linregress(dfz["Length"], dfz["MW"])
>>> lr
LinregressResult(slope=116.18244897959184, intercept=-1871.6131972789153,
rvalue=0.993825553885062, pvalue=1.664932379936294e-22,
stderr=2.765423239336685)
Ce modèle linaire nous indique qu'un résidu a une masse d'environ 116 Dalton, ce qui est cohérent. On peut également comparer ce modèle aux différentes protéines :
>>> plt.clf()
>>> plt.scatter(dfz["Length"], dfz["MW"])
<matplotlib.collections.PathCollection object at 0x7f85b97bfef0>
>>> plt.plot(dfz["Length"], dfz["Length"]*lr.slope+lr.intercept, ls=":")
[<matplotlib.lines.Line2D object at 0x7f85b933e208>]
>>> plt.xlabel("Taille (nombre d'acides aminés)")
Text(0.5, 0, "Taille (nombre d'acides aminés)")
>>> plt.ylabel("Masse moléculaire (Dalton)")
Text(0, 0.5, 'Masse moléculaire (Dalton)')
>>> plt.savefig("transferrine3.png")
On obtient ainsi le graphique de la figure 63.
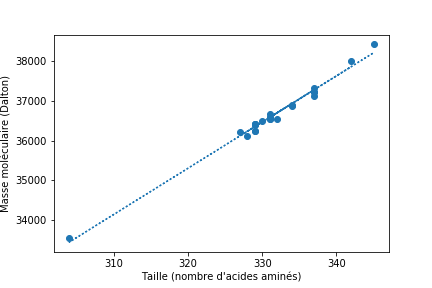
17.5.6 Analyse de données temporelles¶
Il peut être intéressant de savoir, pour chaque organisme, quand les premières et les dernières structures de transferrines ont été déposées dans la PDB.
La méthode .pivot_table() apporte un élément de réponse :
>>> df.pivot_table(index="Source", values=["Deposit Date"], aggfunc=[min, max])
min max
Deposit Date Deposit Date
Source
Anas platyrhynchos 1995-08-03 1996-12-11
Gallus gallus 1993-09-15 2005-09-28
Homo sapiens 1992-02-10 2018-03-22
Oryctolagus cuniculus 1990-08-16 2001-07-24
Sus scrofa 2001-07-03 2001-07-03
Chez l'Homme (Homo sapiens), la première structure de transferrine a été déposée dans la PDB
le 10 février 1992 et la dernière le 22 mars 2018.
Une autre question est de savoir combien de structures de transferrines ont été déposées en fonction du temps.
La méthode .value_counts() peut être utilisée mais elle ne renvoie que
le nombre de structures déposées dans la PDB pour un jour donné. Par exemple,
deux structures ont été déposées le 4 septembre 2000.
>>> df["Deposit Date"].value_counts().head()
1999-01-07 2
2000-09-04 2
2002-11-18 2
2003-03-10 1
2001-07-24 1
Name: Deposit Date, dtype: int64
Si on souhaite une réponse plus globale, par exemple, à l'échelle de
l'année, la méthode .resample() calcule le nombre de structures déposées par
année (en fournissant l'argument A) :
>>> df["Deposit Date"].value_counts().resample("A").count()
1990-12-31 1
1991-12-31 0
1992-12-31 1
1993-12-31 1
Pour connaître en quelle année le plus de structures ont été déposées dans la PDB,
il faut trier les valeurs obtenus du plus grand au plus petit avec la méthode
.sort_values(). Comme on ne veut connaître que les premières dates
(celles où il y a eu le plus de dépôts), on utilisera également la méthode
.head() :
>>> (df["Deposit Date"].value_counts()
... .resample("A")
... .count()
... .sort_values(ascending=False)
... .head())
2001-12-31 5
2003-12-31 4
1998-12-31 3
1999-12-31 3
2002-12-31 3
Name: Deposit Date, dtype: int64
En 2001, cinq structures de transferrine ont été déposées dans la PDB. La deuxième « meilleure » année est 2003 avec quatre structures.
Toutes ces méthodes, enchaînées les unes à la suite des autres, peuvent vous
sembler complexes mais chacune d'elles correspond à une étape particulière
du traitement des données. L'utilisation des parenthèses (ligne 1, juste avant
df["Deposit Date"] et ligne 5, juste après head()) permet de répartir élégamment
cette longue instruction sur plusieurs lignes.
Bien sûr, on aurait pu créer des variables intermédiaires pour chaque étape mais cela aurait été plus lourd :
>>> date1 = df["Deposit Date"].value_counts()
>>> date2 = date1.resample("A")
>>> date3 = date2.count()
>>> date4 = date3.sort_values(ascending=False)
>>> date4.head()
2001-12-31 5
2003-12-31 4
1998-12-31 3
1999-12-31 3
2002-12-31 3
Name: Deposit Date, dtype: int64
- Les ouvrages Python for Data Analysis de Wes McKinney et Pandas Cookbook de Theodore Petrou sont d'excellentes références pour pandas.
17.6 Exercices¶
17.6.1 Nombres pairs et impairs¶
Soit impairs un array NumPy qui contient les nombres 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21. En une seule instruction, construisez l'array pairs dans lequel tous les éléments de impairs sont incrémentés de 1.
Comparez ce que venez de faire avec l'exercice 5.4.4 du chapitre 4 Boucles et comparaisons.
17.6.2 Distance entre deux atomes carbones alpha consécutifs de la barstar¶
La barstar est un inhibiteur de ribonucléase. C'est une protéine relativement simple qui contient 89 acides aminés. Sa structure tridimensionnelle, obtenue par cristallographie aux rayons X, se trouve dans la Protein Data Bank (PDB) sous le code 1BTA.
L'objectif de cet exercice est de calculer la distance entre carbones alpha consécutifs le long de la chaîne peptidique avec module NumPy.
Extraction des coordonnées atomiques¶
Téléchargez le fichier 1bta.pdb qui correspond à la structure de la barstar sur le site de la PDB (lien direct vers le fichier).
Voici le code pour extraire les coordonnées atomiques des carbones alpha de la barstar :
with open("1bta.pdb", "r") as f_pdb, open("1bta_CA.txt", "w") as f_CA:
for ligne in f_pdb:
if ligne.startswith("ATOM") and ligne[12:16].strip() == "CA":
x = ligne[30:38]
y = ligne[38:46]
z = ligne[46:54]
f_CA.write(f"{x} {y} {z} ")
Ligne 1. On ouvre deux fichiers simultanément. Ici, le fichier 1bta.pdb est ouvert en lecture (r)
et le fichier 1bta_CA.txt est ouvert en écriture (w).
Pour chaque ligne du fichier PDB (ligne 2), si la ligne débute par ATOM et le nom de l'atome est CA (ligne 3),
alors on extrait les coordonnées atomiques (lignes 4 à 6) et on les écrit dans le fichier 1bta_CA.txt (ligne 7). Les coordonnées sont toutes enregistrées sur une seule ligne, les unes après les autres.
Lecture des coordonnées¶
Ouvrez le fichier 1bta_CA.txt avec Python et créez une liste contenant toutes les coordonnées sous forme de floats avec les fonctions split() et float().
Affichez à l'écran le nombre total de coordonnées.
Construction de la matrice de coordonnées¶
En ouvrant dans un éditeur de texte le fichier 1bta.pdb, trouvez le nombre d'acides aminés qui constituent la barstar.
Avec la fonction array() du module NumPy, convertissez la liste de coordonnées en array. Avec la fonction reshape() de NumPy, construisez ensuite une matrice à deux dimensions contenant les coordonnées des carbones alpha de la barstar. Affichez les dimensions de cette matrice.
Calcul de la distance¶
Créez maintenant une matrice qui contient les coordonnées des \(n-1\) premiers carbones alpha et une autre qui contient les coordonnées des \(n-1\) derniers carbones alpha. Affichez les dimensions des matrices pour vérification.
En utilisant les opérateurs mathématiques habituels (-, +, **2) et les fonctions sqrt() et sum() du module NumPy, calculez la distance entre les atomes \(n\) et \(n+1\).
Pour chaque atome, affichez le numéro de l'atome et la distance entre carbones alpha consécutifs avec un chiffres après la virgule. Repérez la valeur surprenante.
17.6.3 Jour le plus chaud¶
Le fichier temperature.dat contient un relevé de 4 températures pour chaque jour de la semaine :
À l'aide du module NumPy, on souhaite déterminer quel est le jour de la semaine le plus chaud. Pour cela nous vous proposons les étapes suivantes :
- Récupérez le nom des jours de la semaine depuis le fichier et mettez-les dans une liste
days. - Récupérez les valeurs de températures depuis le fichier et mettez-les dans un array 2D. La fonction
np.loadtxt()et son argumentusecolsvous seront utiles. - Parcourez chaque ligne de la matrice, calculez la température moyenne de chaque jour puis stockez-la dans une liste
mean_temps. - À l'aide des deux listes
daysetmean_temps, déterminez et affichez le jour le plus chaud.
17.6.4 Calcul du centre de masse d'une membrane¶
L'image de gauche de la figure 8 montre le cliché d'une membrane de POPC (cyan) entourée d'eau (bleu) (coordonnées trouvées ici). Les atomes de phosphore des groupes phosphates sont représentés en boule de van der Waals brune. Dans cet exercice on cherche à calculer le centre de masse de la membrane, ainsi que le centre de masse (COM) de chaque monocouche de phosphores. Ces COM sont représentés sous forme de croix dans l'image de droite de la figure 8.
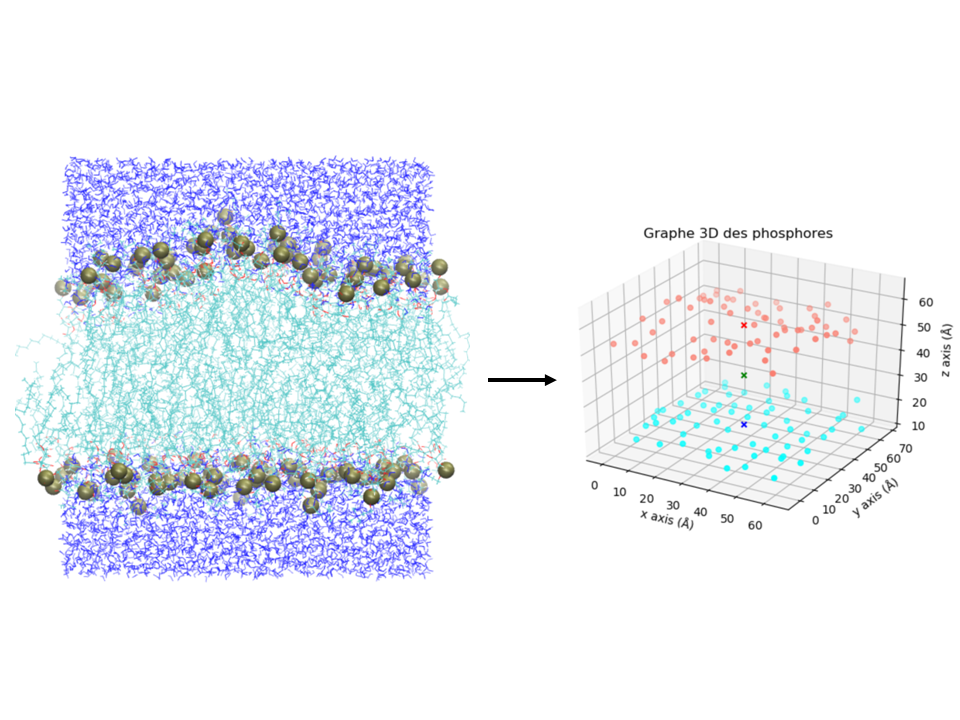
Les coordonnées cartésiennes \((x, y, z)\) de chaque phosphore (en Å) sont stockées dans le fichier coors_P.dat, à raison d'un atome par ligne. Nous vous proposons les étapes suivantes pour résoudre cet exercice à l'aide du module NumPy :
- Récupérez les coordonnées des atomes de phosphore depuis le fichier
coors_P.datet stockez-les dans un array 2D (matrice)coors_P. La dimensionnalité de cette matrice est \(n \times 3\), avec \(n\) le nombre de phosphores. - Calculez le \(z\) moyen de tous les phosphores (nombre réel) et stockez-le dans la variable
mean_z. La méthode.mean()vous sera utile. - Avec des masques de booléens, récupérez les coordonnées des phosphores de la monocouche du haut dans un array 2D
upper. Faites de même avec la monocouche du bas dans un array 2Dlower. - Calculez le centre de masse
COMde la membrane, ainsi que de la monocouche du hautCOM_upperet du basCOM_lower. Pensez aux méthodes de calcul sur les arrays et l'argumentaxis. - Une fois tout cela effectué, créez un graphique 3D pour représenter les différents centres de masse. Vous pourrez utiliser la fonction
scatter()du module matplotlib. Pour l'affichage en 3D, voici un squelette de programme :
# Init plot.
from mpl_toolkits.mplot3d import Axes3D
import matplotlib.pyplot as plt
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
[...]
# X, Y et Z sont des arrays 1D de n éléments (par exemple X représente tous les x des P de la monocouche upper).
ax.scatter(X, Y, Z, c="salmon", marker="o")
# x, y, z sont des floats avec les coordonnées du COM de la upper.
ax.scatter(x, y, z, c="red", marker="x")
[...]
# Axis + title.
ax.set_xlabel("x (Å)")
ax.set_ylabel("y (Å)")
ax.set_zlabel("z (Å)")
ax.set_title("Graphe 3D des phosphores")
plt.show()
17.6.5 Années de publication des articles relatifs à la barstar¶
L'objectif de cet exercice est d'interroger automatiquement la base de données bibliographique PubMed pour déterminer le nombre d'articles relatifs à la barstar publiés chaque année.
Vous utiliserez les modules Biopython et matplotlib.
Requête avec un mot-clé¶
Sur le site de PubMed, cherchez combien d'articles scientifiques sont relatifs à la barstar.
Effectuez la même chose avec Python et la méthode Entrez.esearch() de Biopython.
Choisissez un des PMID renvoyé et vérifiez dans PubMed que l'article associé est bien à propos de la barstar. Pour cela, indiquez le PMID choisi dans la barre de recherche de PubMed et cliquez sur Search. Attention, l'association n'est pas toujours évidente. Cherchez éventuellement dans le résumé de l'article si besoin.
Est-ce que le nombre total d'articles trouvés est cohérent avec celui obtenu sur le site de PubMed ?
Récupération des informations d'une publication¶
Récupérez les informations de la publication dont le PMID est 29701945. Vous utiliserez la méthode Entrez.esummary().
Affichez le titre, le DOI, le nom du journal (Source) et la date de publication (PubDate) de cet article. Vérifiez que cela correspond bien à ce que vous avez lu sur PubMed.
Récupération du résumé d'une publication¶
Récupérez le résumé de la publication dont le PMID est 29701945. Vous utiliserez la méthode Entrez.efetch().
Affichez ce résumé. Combien de caractères contient-il ?
Distribution des années de publication des articles relatifs à la barstar¶
En utilisant la méthode Entrez.esearch(), récupérez tous les PMID relatifs à la barstar. Pour cela, pensez à augmenter le paramètre retmax. Vos PMID seront stockés dans la liste pmids sous forme de chaînes de caractères. Vérifiez sur PubMed que vous avez bien récupéré le bon nombre d'articles.
En utilisant maintenant la méthode Entrez.esummary() dans une boucle, récupérez la date de publication de chaque article. Stockez l'année sous forme d'un nombre entier dans la liste years. Cette étape peut prendre une dizaine de minutes, soyez patient. Vous pouvez dans votre boucle afficher un message qui indique où vous en êtes dans la récupération des articles.
À la fin vérifiez que votre liste years contient bien autant d'éléments que la liste pmids.
Calculez maintenant le nombre de publications par année. Vous créerez pour cela un dictionnaire freq qui aura pour clé les années (oui, une clé de dictionnaire peut aussi être un entier) et pour valeur le nombre de publications associées à une année donnée.
Créez une liste x qui contient les clés du dictionnaire freq. Ordonnez les valeurs dans x avec la méthode .sort(). Créez maintenant une seconde liste y qui contient, dans l'ordre, le nombre de publications associées à chaque années. Bien évidemment, les listes x et y doivent avoir la même taille. Au fait, en quelle année la barstar apparaît pour la première fois dans une publication scientifique ?
Ensuite, avec le module matplotlib, vous allez pouvoir afficher la distribution des publications en fonction des années :
Vous pouvez également ajouter un peu de cosmétique et enregistrer le graphique sur votre disque dur :
import matplotlib.pyplot as plt
plt.bar(x, y)
# redéfinition des valeurs affichées sur l'axe des ordonnées
plt.yticks(list(range(0, max(y), 2)))
# étiquetage des axes et du graphique
plt.xlabel("Années")
plt.ylabel("Nombre de publications")
plt.title("Distribution des publications qui mentionnent la barstar")
# enregistrement sur le disque
plt.savefig("distribution_barstar_annee.png", bbox_inches='tight', dpi=200)
17.6.6 Analyse d'un jeu de données avec pandas¶
Le jeu de données people.tsv contient les caractéristiques de quelques individus : prénom, sexe, taille (en cm) et âge (en années).
Par exemple :
| name | sex | size | age |
|---|---|---|---|
| simon | male | 175 | 33 |
| clara | female | 167 | 45 |
| serge | male | 181 | 44 |
| claire | female | 174 | 31 |
| ... | ... | ... | ... |
L'objectif de cet exercice est de manipuler ces données avec pandas.
Si vous n'êtes pas familier avec le format de fichier .tsv, nous vous conseillons de consulter l'annexe A Quelques formats de données rencontrés en biologie.
Chargement du jeu de données¶
Téléchargez le fichier people.tsv.
Ouvrez ce fichier avec pandas et la fonction .read_csv(). N'oubliez pas de préciser le séparateur par défaut avec l'argument sep="\t". Utilisez également l'argument index_col pour utiliser la colonne name comme index.
Affichez les 6 premières lignes du jeu de données.
Combien de lignes contient le jeu de données ?
Sélections¶
Déterminez la taille de Claire.
Déterminez l'âge de Baptiste.
Affichez, en une seule commande, l'âge de Paul et Bob.
Statistiques descriptives et table de comptage¶
Déterminez la moyenne et la valeur minimale de la taille et l'âge des individus.
Comptez ensuite le nombre de personnes de chaque sexe.
Statistiques par groupe¶
Déterminez, la taille et l'âge moyen chez les hommes et les femmes. Utilisez pour cela la méthode .groupby().
Sélections par filtre¶
Déterminez combien de d'individus mesurent plus de 1,80 m.
Quelle femme a moins de 35 ans ?
Sélections et statistiques¶
Déterminez l'âge moyen des individus qui mesurent plus de 1,80 m.
Déterminez la taille maximale des femmes qui ont plus de 35 ans.